近日,中国科学院上海药物研究所郑明月团队开发出基于生成式AI的蛋白-配体复合物结构预测方法SurfDock。这一方法利用蛋白质表面信息构建几何扩散神经网络,高精度自动生成配体结合构象,并应用于基于结构的虚拟筛选。相关研究成果以SurfDock is a surface-informed diffusion generative model for reliable and accurate protein–ligand complex prediction为题,在线发表在《自然-方法》(Nature Methods)上。
研究蛋白-配体相互作用是分子生物学和生物化学的基础。从酶催化到信号传导,这些相互作用构成了众多细胞过程的核心。研究蛋白-配体相互作用对基于结构的药物设计至关重要,科研人员可据此发现或设计与特定蛋白质结合的配体。科学家利用AI技术能够更精准地剖析蛋白-配体相互作用,加速药物发现进程。
该研究提出了新型基于蛋白表面的几何扩散网络,用于生成精准可靠的蛋白-配体复合物构象。这一模型将多种蛋白质信息如表面特征、残基结构特征和预训练序列特征整合到表面节点的表示中,配备SurfScore内部评分模块,通过对蛋白-配体复合物的训练来评估构象的置信度。同时,SurfDock整合了可选的基于力场的优化步骤,进一步提升了性能。这些设计使得SurfDock在多个基准测试中展现出优异的对接能力,其生成构象的合理性超越现有的深度学习方法。
SurfDock能够适应新的蛋白质、口袋和空结构,在处理高度柔性的配体时表现出色。在实际应用中,研究通过针对ALDH1B1的筛选实验证实了SurfDock的实用价值,快速筛选出7个具有新骨架的先导分子。
研究工作得到国家自然科学基金、国家重点研发计划、中国科学院青年创新促进会会员项目等的支持。
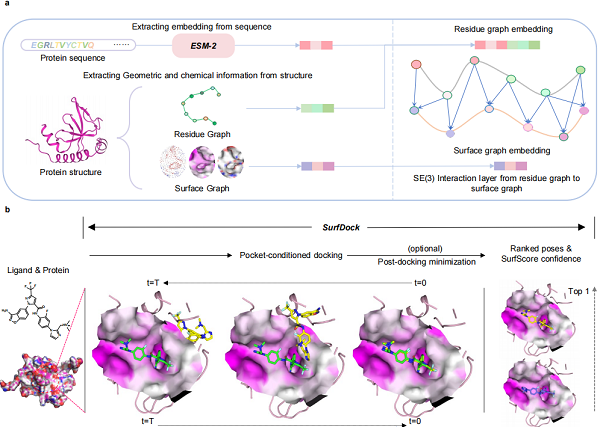
SurfDock架构图